AI&HPC Convergence Frontline

|
COVID-19対策を広げるParabricksによる超高速全ゲノム解析時代の到来
国立大学法人東京大学医科学研究所ヒトゲノム解析センターは、GPUを搭載した最新型のヒトゲノム解析用スーパーコンピュータシステムSHIROKANE (以下、SHIROKANE)に、GPUサーバ(DGX A100)を新たに加えた全88基のGPUサーバに、NVIDIA Clara™ Parabricks(以下、Parabricks)を全面導入し、スケジューラやストレージ等の既存システムとの連携を考慮し最適化を図った。(プレスリリース) 2021年3月1日に稼働を開始し、これまでの処理能力が16基から88基の約6倍もの拡張となり、さらにParabricksによる処理能力の向上により、SHIROKANEの全ゲノム解析のさらなる高速化が期待できるものとなった。

今回、全GPUサーバでの利用に拡大されたParabricksは、NVIDIAが提供するヘルスケア領域に位置するソフトウェアの1つで、ゲノム解析における共通した解析処理に対して、高度にGPUに最適化されたCUDA MapperやCUDA Aligner、CUDA POAのライブラリを提供するツールキットとして構成されている。そして、提供されるParabricksパイプラインとしては、マサシューセッツ工科大学とハーバード大学が共同で運営するブロード研究所が開発したGenome Analysis Toolkit (GATK)のバージョン4.1を利用する事ができる。
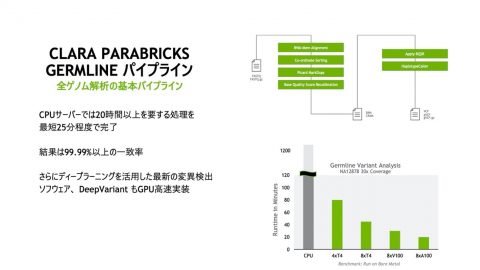
現在1800人以上のSIROKANEを利用するユーザの中でも、全ゲノム解析環境の高速化は最前線の臨床ゲノム研究者が望んでいたもので、国内最大規模の生命・医学系専用のスパコンであるSHIROKANEにParabricksが全面採用された意義は大きい。一般的なCPU環境では1サンプルあたり20時間以上を要する処理が、DGX A100 (GPU 8基)での計測では、30分以内に完結する事が出来きた。これは実に従来の約40倍の高速化となる。もちろん、SHIROKANEに搭載されたGPUサーバの多くは、Tesla V100であるが、全てのGPUを使って利用できるようになった事で、全ゲノムデータ解析を実現する基盤強化が最大限図られた事になる。
 |
ヒトゲノム解析センターセンター長、井本清哉教授によると、同センターでは、医科研病院と先端医療研究センターとの共同研究で、SHIROKANE上に構築されたParabricks環境を使った解析を、がん治療の臨床現場にも利用するとの事だ。その現場では、医科研病院のガン患者から同意のもとに提供されるゲノムの変異を調べ、その変異を臨床的に解釈した情報を主治医に提供し、投薬の効果や予後の改善に活用していく。
 |
|
特に病態の進展の早い急性骨髄性白血病の場合には、1週間といった短い期間に、提供された生体サンプルから、ゲノムシーケンス解析やデータ解析といった一連の処理を経て、治療計画を提供する必要があり、これまで10時間以上掛かっていた計算をParabricksにより100分に短縮できる知見を得たと説明する。そして、ゲノム医療の近未来は、現在の標準治療を終了した患者を対象としたゲノム上の数百の遺伝子の状態を調べるパネル検査ではなく、全ゲノムシーケンスと人工知能(AI)による解析が、がんや先天性の疾患や代謝異常などに対して利用が広がるとの見解を示している。
実際に1年間にがんと診断される数は100万人に近い現在においては、生涯がんに罹患する確率は、実に50%以上あるとされることから、全ゲノムシーケンスのデータ解析の潜在的ニーズは高いといえるだろう。だからこそ、大量の計算処理と、大量のファイルアクセスが発生する全ゲノム解析を、実際の臨床の場で定常的に動かすITエンジニアリング的な知見の蓄積は、今後この手の解析を広げて行くためには重要となる。
さらに、この直近の1年に限って言えば、国際的には、新型コロナウィルス感染症(COVID-19)に関連して、早い段階で感染者のゲノムシーケンスデータを解析する国際コンソーシアムが立ち上がり、感染者の重症化の度合いと、遺伝子変異との関係について調査が行われており、全ゲノムシーケンスの重要性は、さらに増加していくとのことだ。日本国内の動きとしては、コロナ制圧タスクフォースが立ち上がり、参画する東京大学医科学研究所は、日本人やアジア人の集団における遺伝的背景が、重症化率や死亡率にあたえる関係を、ゲノム上のSNPや全ゲノム解析、RNA解析するゲノム情報学(データサイエンス)の視点でアプローチする。
COVID-19の制圧には、新型コロナウィルス自体のゲノムの情報に加え、ヒト免疫反応に関連する全ての情報を含むビックデータの解析が不可欠と考えられており、Parabricksを活用できるGPUサーバの拡大と増強は、急激に増える解析ニーズに対応した形となる。Parabricksを利用するジョブについては、1台のサーバ筐体にある8基のGPUを使うのがベストプラクティスとなっているようで、これまでSHIROKANEは、ライセンスの関係で2個のParabricksジョブを同時に実行できていたが、今回全てのGPUで利用できるライセンスを入手したことから、11個のジョブを同時に実行できるようになった。そのライセンスの追加に関連して、各種SHIROKANEの基盤システムも最適化されたようだ。
 |
今後、全ゲノム解析により取得されたパーソナルゲノム情報に基づいた予防・診断・治療法についての注目される中、今回のParabricks導入による東京大学医科学研究所ヒトゲノム解析センターの成果や知見は、関連業界が注目するものとなるだろう。なお、医科学研究所では、COVID-19に関する疫学・バイオインフォマティクス・分子モデリングなどの研究プロジェクトに関して、無償で一定数の計算資源を提供しており、Parabricksを利用できるGPUサーバも含まれているため、関連研究の促進に期待がもてる。
Parabricksは、NVIDIAにより有償の年間ソフトウェアライセンスにより提供される製品となっており、価格は非公開となっている。また、A100に類するGPUを持つサーバ1台から利用する事ができ、大学・研究所だけでなく、企業での導入実績もあるそうだ。
用語について:
全ゲノム解析 生物のゲノムのもつ遺伝情報を総合的に解析すること
SNP(スニップ) 個人間の遺伝情報のわずかな違いのこと。一塩基多型(いちえんきたけい、single nucleotide polymorphism)
RNA解析 RNA解析(トランスクリプト―ム解析)とは、次世代シーケンサー(NGS)により全転写物の塩基配列を決定する方法。

